中南大学计算机学院、生物信息学湖南省重点实验室王建新教授和昆明理工大学信息工程与自动化学院、云南省计算机技术应用重点实验室彭玮教授在抗癌药物反应预测领域取得重要研究成果。该研究成果以“Predicting cancer drug response using parallel heterogeneous graph convolutional networks with neighborhood interactions” 为题,在国际生物信息学权威期刊《Bioinformatics》(IF=6.931)上在线发表。
https://doi.org/10.1093/bioinformatics/btac574
由于癌症的异质性,当一组相同癌症类型的患者接受相同治疗时,治疗效果可能不同。抗癌药物反应预测有助于开发个性化治疗方案,以提高生存率并减少患者的费用。最近,基于图神经网络的方法引起了广泛的兴趣,并在药物反应预测任务上取得了令人瞩目的成果。然而,他们中的大多数应用图卷积来处理细胞系-药物二部图,而忽略了细胞系和药物节点之间的内在差异。此外,这些方法中的大多数都聚合了节点邻域特征,但没有考虑细胞系和药物之间的元素交互。课题组提出了一种基于邻域交互的异构图卷积网络方法,即 NIHGCN,用于抗癌药物反应预测。首先,它构建了一个由药物、细胞系和已知的药物反应信息组成的异构网络。细胞系基因表达和药物分子指纹分别进行线性变换并作为节点属性输入到交互模块中。交互模块由并行图卷积网络层和邻域交互层组成,细胞系和药物分别通过图卷积操作聚合来自其邻居的节点级特征,并通过邻域交互层考虑与其邻居元素级的交互。最后,通过计算细胞系和药物最终特征表示的线性相关系数来进行药物反应预测。与最先进的算法相比,NIHGCN在多项实验上取得了最好的性能。在 GDSC 和CCLE数据集上的 AUC 分别达到 0.876 和 0.8806,并成功预测了新细胞系和新药的药物反应。此外,NIHGCN在 GDSC 数据集上训练的模型可以成功地应用于预测 PDX 和 TCGA 的样本,这验证了NIHGCN从体外细胞系到体内数据集的可移植性。
王建新教授团队长期致力于计算机算法与优化、生物信息学、医学影像分析等方面研究。彭玮教授团队长期致力于生物信息学、数据挖掘等方面研究。该研究受到国家自然科学基金、国家自然科学基金委员会-浙江省人民政府两化融合联合基金、云南省应用基础研究计划、云南省兴滇人才计划等基金资助。
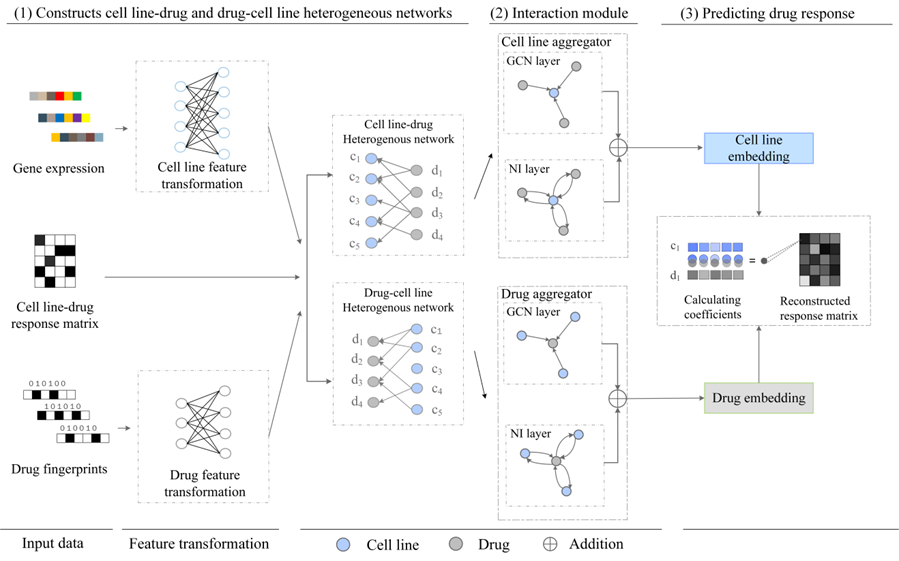
NIHGCN模型的框架图