中南大学计算机学院、生物信息学湖南省重点实验室成员2019级博士张俊在第三代序列纠错领域取得重要研究成果。该研究成果以Fec: a fast error correction method based on two-rounds overlapping and caching为题,在国际生物信息学权威期刊《Bioinformatics》(IF=6.937)上在线发表。

第三代测序技术以其长读长的特点加速了基因组分析,但其高错误率的特点会给下游分析带来偏差,因此纠错是一个很重要的步骤。纠错是一个非常耗时的过程,尤其是在测序深度较高的时候。在对具有重叠关系的两条读数纠错时,现有基于POA的纠错方法需要两次碱基级别的比对。然而,这两次的比对结果是可以相互推断出来的。课题组提出并开发了一种基于两轮比对和缓存的快速纠错方法Fec。Fec既可以作为纠错工具单独使用,也可以作为组装流程的纠错步骤使用。在第一轮比对中,Fec快速地找到足够多的重叠关系来纠错大部分的读数。在第二轮比对中,Fec为在第一轮比对中找到的重叠关系不足以纠错的读数找到尽可能多的重叠关系。在对具有重叠关系的两条读数进行碱基级别的比对时,Fec首先搜索缓存,如果缓存中存在这两条读数的比对结果,就将其提取出来并根据这个比对结果推断出第二个比对结果。否则,Fec就进行碱基级别的比对并将比对结果保存到缓存中。与现有的多种纠错方法对比,Fec实现了1.16-38.56倍的加速且准确率相同甚至更高。
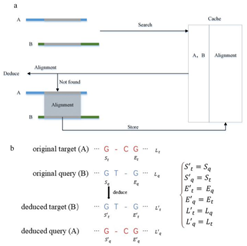
缓存和比对结果推断图
王建新教授团队长期致力于计算机算法与优化、生物信息学、医学影像分析等方面研究。该研究工作得到国家重点研发计划、国家自然科学等基金支持。