中南大学计算机学院、生物信息学湖南省重点实验室成员郭林沅在蛋白质分子对接领域取得重要研究成果。该研究成果以“TRScore: a 3D RepVGG-based scoring method for ranking protein docking models”为题,在国际生物信息学权威期刊《Bioinformatics》(IF=6.937)上在线发表。

蛋白质分子对接在蛋白质功能研究与生物医药研发领域中是不可或缺的研究手段。目前蛋白质分子对接的主要问题在于如何设计一个打分函数,能更有效地从计算方法搜索出的大量蛋白-蛋白结合模式(decoys)中筛选出近天然的模式(near-native decoys)。其中主要的评价指标为success rate、hit rate和score-RMSD相关系数。该研究成果开发了一种名为TRScore的算法,用于对大量蛋白-蛋白结合模型进行打分,使得近天然的结合模式能尽量根据打分值排在其它模式前面。亲疏水性在蛋白质相互作用中起到至关重要的作用,TRScore使用了19种基于亲疏水性的蛋白质原子分类,并把蛋白-蛋白相互作用界面栅格化,从而在每一个栅格中填入19原子分类的向量。立体栅格建立后,TRScore使用RepVGG作为三维卷积神经网络的架构,从界面提取相互作用特征,然后输入到全连接神经网络,最后得出该结合模式的打分值。实验结果表明,TRScore在ZDOCK benchmark5.0、DockGround、CAPRI数据上超越了现有的方法,在success rate、hit rate和score-RMSD相关系数取得最好结果。

(a). 立体栅格建立过程
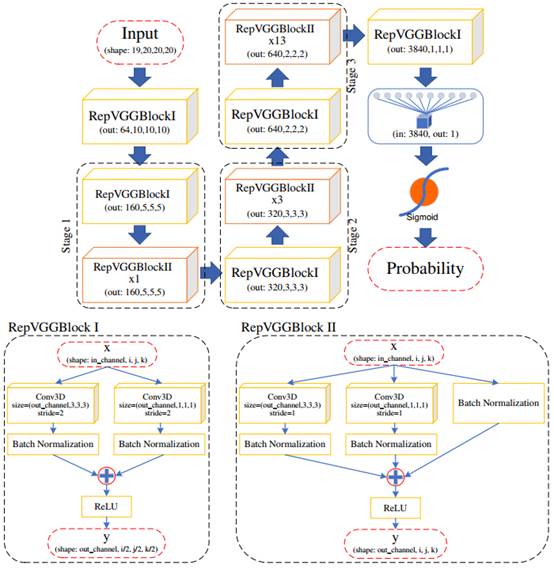
(b). RepVGG三维卷积神经网络结构