中南大学计算机学院、生物信息学湖南省重点实验室成员黄能在第三代牛津纳米孔测序领域取得重要研究成果。该研究成果以“BlockPolish: accurate polishing of long-read assembly via block divide-and-conquer”为题,在国际生物信息学权威期刊《Briefings in Bioinformatics》(IF=11.622)上在线发表。

Nanopore测序具有错误率高(7%)、错误分布广泛(3-30%)和局部错误不均的特点,这使得基因组组装结果中不同区域的错误率和错误分布存在极大的差异。在对基因组组装结果进行纠错时,现有的算法并未考虑到基因组不同区域的错误分布的差异,而是将不同区域同等看待。BlockPolish通过将基因组划分成块,统计每个块的错误率以及错误分布,区分出低复杂度区域和高复杂度区域。在低复杂度区域基因组组装结果的错误率较低,错误分布集中为替换错误和删除错误;而高复杂度区域的错误率较高,错误分布除了替换和删除错误,还包括插入错误。对于低复杂度区域,BlockPolish使用深度学习模型根据每个位置的碱基数量预测纠错序列。对于高复杂度区域,由于存在大量的插入错误,使得该区域上序列比对结果较差,因此BlockPolish利用多序列比对算法对高复杂度区域的长读数进行重新对齐,优化比对结果。然后利用深度学习模型从重新对齐的比对结果预测纠错序列。在深度学习结构设计方面,BlockPolish采用多任务框架,一个任务预测压缩的纠错序列,目的是让模型更加关注纠错序列上不同碱基出现的次序;另一个任务是预测全长的纠错序列,全长的纠错序列不光包含不同碱基的出现次序信息,还有每个碱基在某个位置上重复的次数。由于测序技术的限制,精准地确定碱基重复次数在纳米孔组装纠错上是一个难题,BlockPolish在全长序列预测中借助Flip-flop的思想,区别连续重复的碱基,从而达到提高预测精度的目的。
BlockPolish与现有的多种抛光算法Racon、Medaka、MarginPolish & HELEN进行比较,在全基因组人类数据上,BlockPolish具有最高的抛光精度、最佳的基因完整度;同时BlockPolish对不同组装算法产生的组装结果、不同精度测序数据产生的组装结果都有非常好的适应性和鲁棒性。
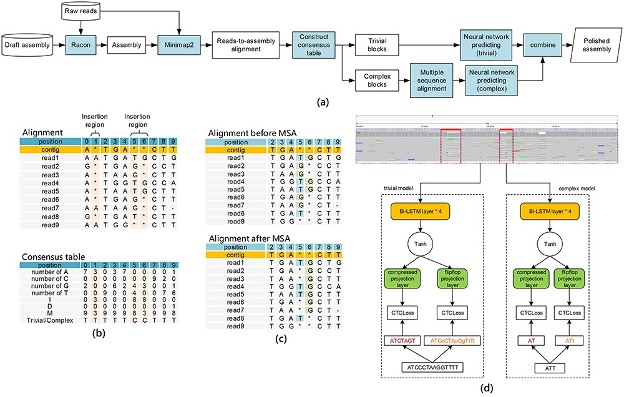
(a)算法主要流程。(b)构造一致性表划分低复杂度区域和高复杂度区域。
(c)多序列比对重新对齐高复杂度区域,优化比对结果。(d)神经网络模型结构。