中南大学计算机学院、生物信息学湖南省重点实验室成员黄能在第三代牛津纳米孔测序领域取得重要研究成果。该研究成果以“NeuralPolish: a novel Nanopore polishing method based on alignment matrix construction and orthogonal Bi-GRU Networks”为题,在国际生物信息学权威期刊《Bioinformatics》(IF=5.61)上在线发表。

纳米孔测序以低成本产生超长的reads,在基因组学研究中取得了许多突破。然而,纳米孔测序组装结果中的大量错误影响了基因组分析的准确性。polishing是一个纠正组装中的错误的过程,可以提高下游分析的可靠性。该方法从reads-to-assembly的比对结果中编码并构造特征矩阵,极大程度减少从比对结果转换成神经网络输入的信息损失。同时利用一个正交双向GRU网络对分别对比对矩阵进行按行、按列计算上下文关系。按行处理目的是计算单条read序列上下文信息,同时减小多条reads之间的相互影响。按列处理目的是根据多条reads的碱基计算染色体上每个位置的不同碱基的概率。最终由CTC解码器根据每个位置的碱基概率得到纠错后的组装序列。NeuralPolish与现有的多种polishing算法Racon、MarginPolish、HELEN和Medaka在不同组装工具Wtdbg2、Flye和Canu上进行对比,纠错效果提升明显,纠错后的组装结果精度更高。
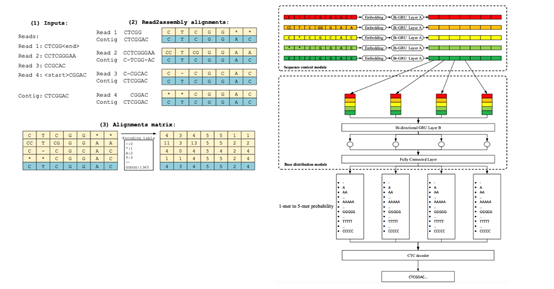
NeuralPolish核心方法:比对矩阵构造和正交双向GRU网络