中南大学计算机学院、生物信息学湖南省重点实验室成员黄能在第三代牛津纳米孔测序领域取得重要研究成果。该研究成果以“SACall: a neural network basecaller for Oxford Nanopore sequencing data based on self-attention mechanism”为题,在国际生物信息学权威期刊《IEEE/ACM TRANSACTIONS ON COMPUTATIONAL BIOLOGY AND BIOINFORMATICS》(IF=3.015)在线发表。

第三代牛津纳米孔测序仪的因其便携性,低成本,实时测序等优势,在基因组学研究方面取得了许多突破。但是,相比于第二代短读数测序99%的准确率,纳米孔测序的主要缺陷是它的高错误率。尽管近几年纳米孔测序的化学试剂和basecalling工具在不断地更新和发展,但是测序的错误率仍然在5%~15%。极高的错误率使得纳米孔测序在许多下游分析中存在诸多局限性和不可靠性。该研究提出了一种新型的端到端的basecalling方法SACall。在模型中组合了卷积神经网络,Transformer自注意力网络。多层卷积网络用于下采样测序信号并捕获局部模式。自注意力网络用于计算原始信号序列中任意两个位置上信号的上下文关系。最后,采用束搜索的方法计算最终的DNA序列。该研究在多个标准测试数据上对SACall和纳米孔测序公司官方工具Albacore,Guppy进行评估。SACall从reads准确度,基因组组装质量和共识序列准确度等多个方面都要比官方basecalling工具有明显提升。该研究为提高纳米孔测序精度提供了新的方法。
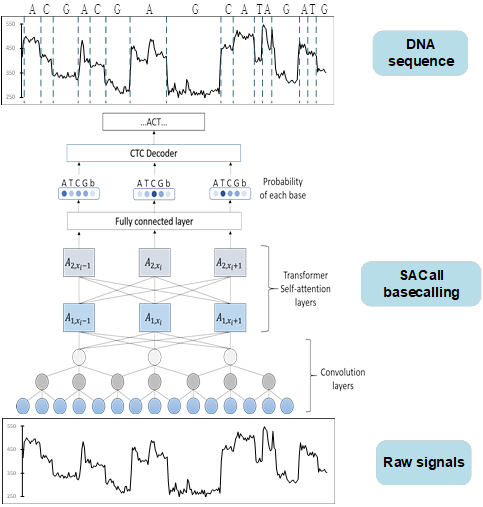
Nanopore原始测序信号翻译为DNA核苷酸序列的主要流程