中南大学计算机学院、生物信息学湖南省重点实验室成员杨梦云在计算药物重定位领域取得重要研究成果。该研究成果以“Computational Drug Repositioning Based on Multi-Similarities Bilinear Matrix Factorization”为题,被国际生物信息学权威期刊《Briefings in Bioinformatics》(IF=8.99)录用。中南大学计算机学院、生物信息学湖南省重点实验室博士杨梦云为论文的第一作者,王建新教授为论文的通信作者,中南大学为第一单位。

为了有效地融合药物和疾病的多种相似性数据,我们提出基于多相似性矩阵分解(MSBMF)方法来预测现有药物和新颖药物的潜在适应症。针对药物(或疾病)的多个相似矩阵,我们不是将多个相似矩阵融合成一个单一的药物(或疾病)相似矩阵,而是将这些相似性矩阵分别进行拼接。相比融合成单一的相似性矩阵,这样的操作能完好地保持各类相似性数据的分布特征,能在优化的过程中全面地获取药物和疾病的特征表示。我们使用矩阵分解方法将药物-疾病关联矩阵用药物潜藏特征和疾病潜藏特征的乘积表示。在建模的过程中,我们使用药物的潜藏特征将药物的多相似性矩阵线性表示出来,类似地,使用疾病的潜藏特征将疾病的多相似性矩阵线性表示出来,这样可以有效地将关联数据和多相似数据一起并入模型优化中。此外,为了确保预测的药物-疾病关联值都是非负的,我们在矩阵分解的目标函数中加入了非负约束,这样可以使得预测值在生物学上具有更好的可解释性。最后,利用交替方向乘子法(ADMM)对MSBMF模型进行了求解。实验结果表明,在交叉验证实验中,MSBMF方法比目前先进的药物重定位方法具有更高的预测精准度。同时,案例分析也证明了MSBMF方法在实际应用中的有效性。
王建新教授团队长期致力于计算机算法与优化、生物信息学、医学影像分析等方面研究。该研究工作得到了国家自然科学基金和湖南省研究生科研创新项目的资助。
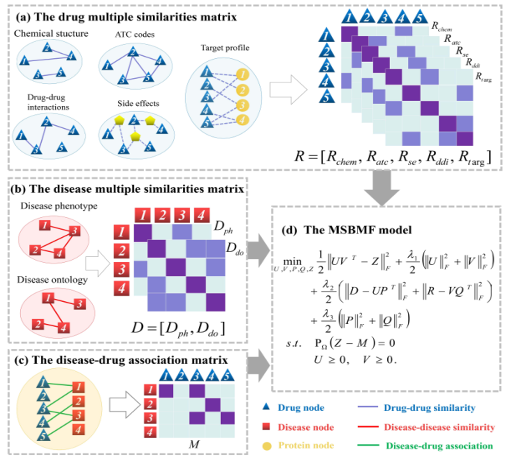
MSBMF模型的流程图