中南大学计算机学院、生物信息学湖南省重点实验室成员倪鹏在三代测序碱基修饰检测领域取得重要研究成果。该研究成果以“RNA m6A detection using raw current signals and basecalling errors from Nanopore direct RNA sequencing reads”为题,在国际生物信息学权威期刊《Bioinformatics》上在线发表。
m6A (N6-methyladenosine) 修饰是RNA中最丰富的碱基修饰之一,约25%的mRNA中存在该修饰。在真核生物RNA中,m6A影响RNA的结构和稳定性,在mRNA翻译、剪接等多种生物学过程中发挥重要作用。由于其独特测序技术,纳米孔直接RNA测序 (DRS) 无需额外的实验室手段即可检测RNA m6A。然而,现有基于纳米孔测序的计算方法通常只利用了测序信号的统计特征或碱基识别特征,忽略了DRS测序读数原始信号的更丰富信息。基于此,课题组提出了一种深度学习方法RedNano,旨在充分利用纳米孔原始测序信号和碱基识别特征从DRS测序读数中检测m6A。RedNano从纳米孔DRS数据中提取原始信号特征和碱基识别特征,然后构建残差网络分别处理这两种特征,实现了读数水平和位点水平的RNA m6A检测。在合成RNA数据、拟南芥和人类数据上的测试结果表明,RedNano获得了比其他方法更高的AUC和AUPR。此外,RedNano在跨物种验证中表现比其他方法更佳,表明其具有较高稳健性。在对毛果杨独立数据集上的测试表明,RedNano同样获得了最高的AUC和AUPR,且其AUC和AUPR分别比其他方法高出3.8%-9.9%和5.5%-13.8%。
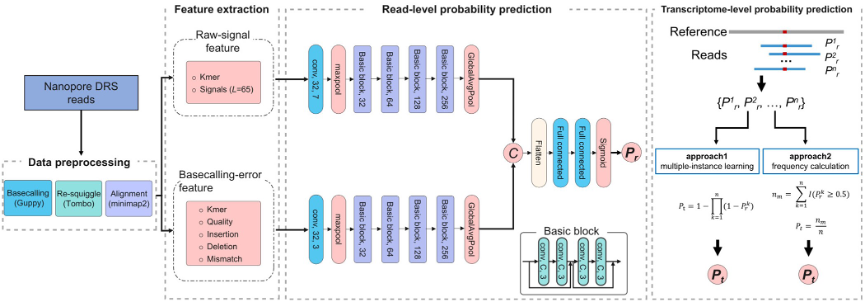
王建新教授团队长期致力于计算机算法与优化、生物信息学、医学影像分析等方面研究。该研究工作得到国家重点研发计划、国家自然科学基金、湘江实验室揭榜挂帅项目以及中南大学高性能计算平台的支持。