中南大学计算机学院、生物信息学湖南省重点实验室成员毕雪华博士在基因与表型关系预测领域取得了重要研究成果。该研究成果以“SSLpheno: A Self-Supervised Learning Approach for Gene-Phenotype Association Prediction Using Protein-Protein Interactions and Gene Ontology Data”为题,在国际生物信息学权威期刊《Bioinformatics》(IF= 5.8)上在线发表。

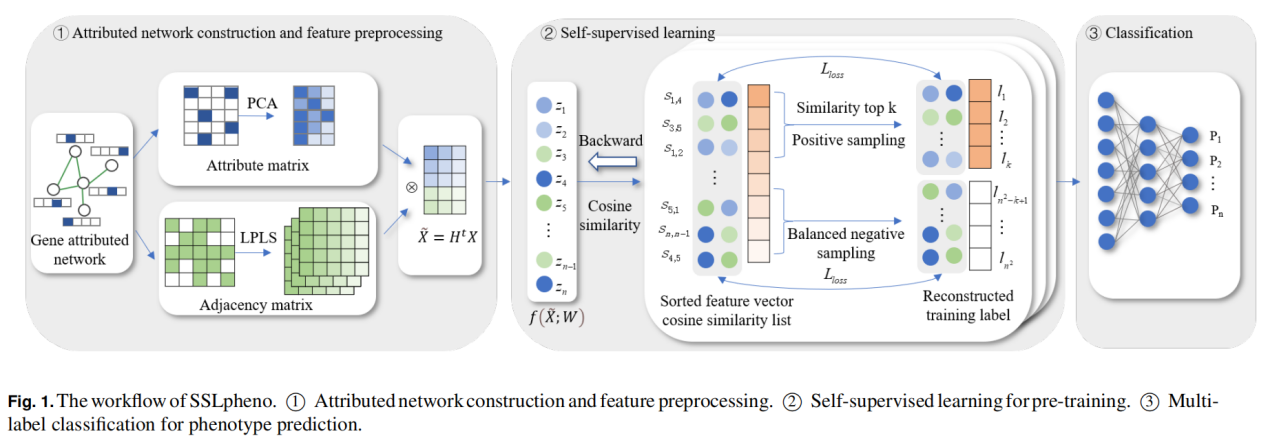
基因与疾病表型的形成密切相关,近年来,发现了大量与疾病表型相关的候选基因。这些发现极大地提高了我们对遗传疾病的理解,以及新药和新药物治疗方法的开发。传统的研究方法,如全基因组关联研究(Genome Wide Association Studies, GWAS),并不能解释所有的疾病风险。随着人类表型计划(Human Phenome Project, HPP)的提出,促进了预测基因-表型关联的计算方法的发展。然而,尽管建立了标准化的疾病表型数据库,预测基因-表型关联的计算方法仍然存在类别分布不平衡和缺乏小类别标记数据的问题。在本文中,作者提出了一种基因-表型关联预测的自监督学习模型,称为SSLpheno。此方法利用了一个整合了蛋白质相互作用和基因本体数据的属性网络,使用一个基于拉普拉斯的滤波器来确保特征的平滑性,并使用自监督训练来优化节点特征表示。在下游任务中,我们采用深度神经网络进行多标签分类。实验结果表明,SSLpheno的性能优于最先进的方法,特别是在注释较少的类别中。此外,案例研究证明了SSLpheno具有基因-表型关联鉴定的有效预筛选工具的潜力。
王建新教授团队长期致力于计算机算法与优化、生物信息学、医学影像分析、医疗数据挖掘等方面研究。该研究工作得到国家重点研发项目、国家自然科学基金等基金支持。