中南大学计算机学院、生物信息学湖南省重点实验室成员张立身博士和卢诚谦博士后在RNA-Protein相互作用领域取得了重要研究成果。该研究成果以“CRMSS: predicting circRNA-RBP binding sites based on multi-scale characterizing sequence and structure features”为题,在国际生物信息学权威期刊《Briefings in Bioinformatics》(IF= 13.994)上在线发表。

https://doi.org/10.1093/bib/bbac530
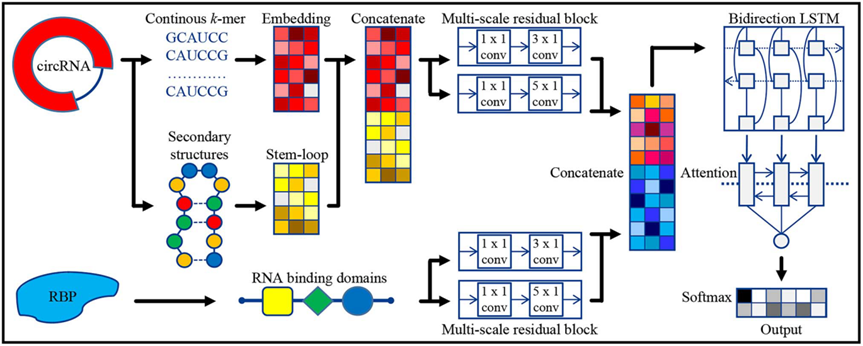
Circular RNAs(circRNAs)是一种类反向剪切并且共价闭合的RNA。有些circRNA可以作为RNA结合蛋白(RBPs)海绵,对多种疾病的发生发展有显著影响,是时下研究的热点。通过实验验证circRNA-RBP相互作用耗时且成本高昂,因此急需一些计算方法预测这种相互作用,减少实验前期准备。已有的计算方法仅使用circRNA的序列信息预测RBP的结合,没有全面考虑circRNA-RBP结合的生物特性。本研究首次同时考虑了circRNA和RBP两方面的序列和结构特征,全面描述这一过程的生物特性。我们提出一种基于多尺度的序列和结构特征识别circRNA-RBP结合位点的预测方法,简写作CRMSS。对于circRNA,我们使用序列的k-mer 嵌入表示和局部二级结构的形成概率作为特征。对于RBP,我们将RBP上RNA结合域的序列单元和结构单元的种类组合起来,生成混合的频率特征。CRMSS采用多尺度的残差模块捕捉特征中的结合模式,之后通过具有注意力机制的BiLSTM学习结合模式的局部和全局表示。在Clip-seq和RIP-seq两种数据上,CRMSS取得了优于已有计算方法的预测性能。来自circRNA和RBP的序列和结构的特征使模型捕获到了可靠的序列基序(motif),显著提高了模型的预测效果。CRMSS还能正确反映RBP对circRNA的结合偏好,对训练数据集外的结合位点做出准确的预测。这些预测结果能够在一定程度上揭示circRNA-RBP相互作用的潜在生物机制,为后续的实验验证提供思路。

王建新教授团队长期致力于计算机算法与优化、生物信息学、医学影像分析、医疗数据挖掘等方面研究。该研究工作得到国家重点研发项目、国家自然科学基金、111计划等基金支持。