中南大学计算机学院、生物信息学湖南省重点实验室成员在剪接异构体功能预测研究领域取得重要研究成果。该研究成果以“IsoResolve: Predicting Splice Isoform Functions by Integrating Gene and Isoform-level Features with Domain Adaptation”为题,在国际生物信息学Top期刊《Bioinformatics》(IF=5.610)上被接收。中南大学计算机学院、生物信息学湖南省重点实验室特聘副教授李洪东和科研助理杨昌获为论文共同第一作者,王建新教授为论文的唯一的通信作者,中南大学为唯一通讯单位。

基因功能的高分辨率注释是功能基因组学的核心目标。一个基因可以通过选择性剪接产生多种功能不同的剪接异构体。然而,传统的方法将基因视为一个单一的实体,不区分其功能不同的剪接异构体。正确、高效的区分同基因中不同剪接异构体的功能对更高分辨率理解基因功能、辅助疾病诊断、制定治疗方案等方面具有非常重要的意义。现有剪接异构体功能预测方法的预测性能远远不能令人满意,其主要原因是由于剪接异构体功能注释的缺乏。该研究成果提出了一种新的剪接异构体功能预测方法IsoResolve。因为基因数据与剪接异构体数据高度相关且基因层面有着丰富的功能注释信息,团队成员提出利用迁移学习的思想将基因功能预测模型加入剪接异构体功能预测,很好地解决了剪接异构体功能注释缺乏的问题,显著提高了预测性能。本研究为更精准地区分剪接异构体功能提供了新的方法。
王建新教授团队长期致力于计算机算法与优化、生物信息学、医学影像分析等方面研究。该研究工作得到国家自然科学、高等学校学科创新引智计划、湖南省科技计划等基金支持。
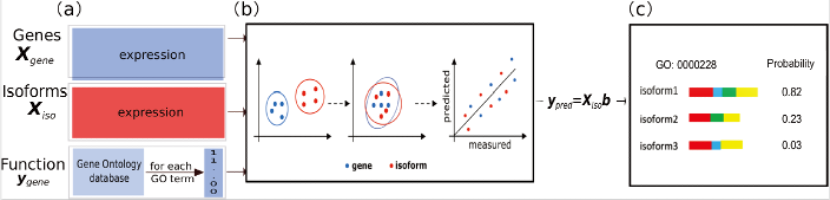
IsoResolve功能预测主要流程