近日,中南大学计算机学院、生物信息学湖南省重点实验室成员成建宏博士在脑胶质瘤分级诊断研究上取得重要研究成果。该研究成果以 “Multimodal Disentangled Variational Autoencoder with Game Theoretic Interpretability for Glioma Grading”为题,在生物医学和健康信息学国际Top期刊《IEEE Journal of Biomedical and Health Informatics》(IF=5.772) 上见刊出版。

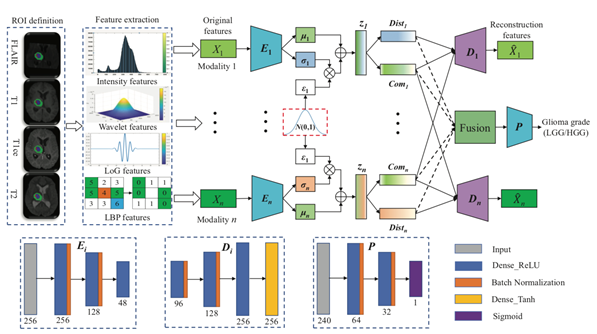
由于不同影像模态可提供互补信息,多模态磁共振影像(MRI)的有效融合对提高胶质瘤分级的准确性具有重要意义。然而,如何从MRI中提取共同信息和特异信息实现信息互补仍然是信息融合研究中的一个开放性问题。为此,课题组提出了基于多模态MRI特征解耦的变分自编码器脑胶质瘤自动分级(MMD-VAE)。为了提取多模态MRI影像间的互补和共享特性,设计了跨模态重构损失和“共享-特异”损失进行特征解耦,并融合这些解耦特征进行脑胶质瘤分级。于此同时,为了提升模型可解释性,课题组采用了一种基于博弈论可解释性方法——SHAP method来定量解释和分析重要特征对分级的贡献度。实验结果表明,所提出的MMD-VAE模型在公共数据集上实现了较高的预测性能(AUC:0.9939),并在湘雅二医院的私有数据集上得到了进一步验证,并显示出良好的泛化性能(AUC:0.9611)。该研究有望可以辅助放射医师更好地理解胶质瘤分级,并作更好的临床决策。
王建新教授团队长期致力于计算机算法与优化、生物信息学、医学影像分析等方面研究。该研究工作得到国家自然科学、湖南省自然科学、湖南省科技计划等基金支持。